如何進行大數據中R語言的缺失值處理
本篇文章給大家分享的是有關如何進行大數據中R語言的缺失值處理,小編覺得挺實用的,因此分享給大家學習,希望大家閱讀完這篇文章后可以有所收獲,話不多說,跟著小編一起來看看吧。
10年積累的成都網站設計、網站建設經驗,可以快速應對客戶對網站的新想法和需求。提供各種問題對應的解決方案。讓選擇我們的客戶得到更好、更有力的網絡服務。我雖然不認識你,你也不認識我。但先建設網站后付款的網站建設流程,更有猇亭免費網站建設讓你可以放心的選擇與我們合作。
拿到數據后,在清楚了分析需求后,別急著各種統計、模型一塊上,先給數據做個“清潔”再說。數據中往往會有各種缺失值,異常值,錯誤值等,今天先介紹一下如何處理缺失值,才能更好的數據分析,更準確高效的建模。
一 查看數據集的缺失情況
R中使用NA代表缺失值,用is.na識別缺失值,返回值為TRUE或FALSE。由于邏輯值TRUE和FALSE分別等價于數值1和0,可用sum()和mean()來獲取數據集的缺失情況。
載入R包及內置數據集
library(VIM)#VIM包的sleep數據集示例data(sleep,package="VIM")
1)查看數據集整體有多少缺失值及百分比
sum(is.na(sleep))mean(is.na(sleep))
2)查看數據集特定變量(列)有多少缺失值及百分比
sum(is.na(sleep$Sleep))mean(is.na(sleep$Sleep))
3)數據集中多個行包含缺失值
mean(!complete.cases(sleep))
4)列出沒有缺失值的行
sleep[complete.cases(sleep),] #利用函數list <-which(rowSums(is.na(sleep)) > 0) ; sleep[-list,] # 效果同上
5)列出有一個或多個缺失值的行
sleep[!complete.cases(sleep),]list <-which(rowSums(is.na(sleep)) > 0) ; sleep[list,]
二 探索缺失值
2.1 mice包展示數據整體的缺失情況
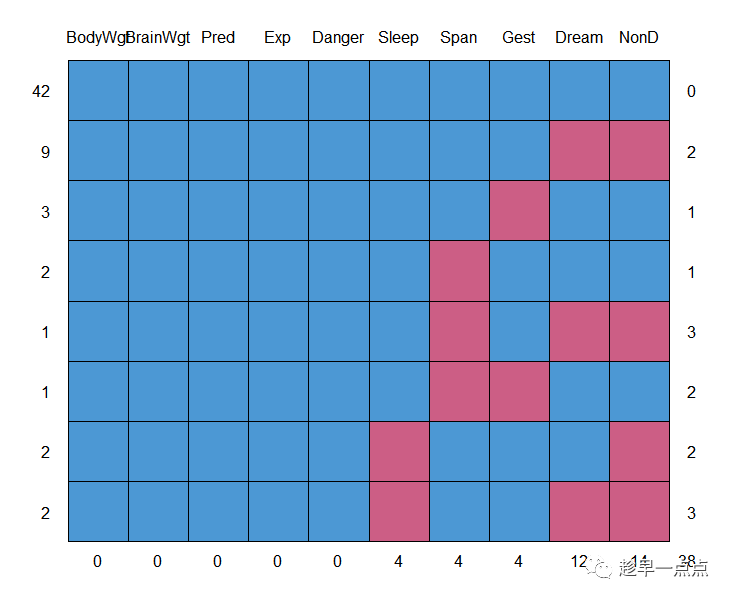
library(mice)md.pattern(sleep)BodyWgt BrainWgt Pred Exp Danger Sleep Span Gest Dream NonD 42 1 1 1 1 1 1 1 1 1 1 09 1 1 1 1 1 1 1 1 0 0 23 1 1 1 1 1 1 1 0 1 1 12 1 1 1 1 1 1 0 1 1 1 11 1 1 1 1 1 1 0 1 0 0 31 1 1 1 1 1 1 0 0 1 1 22 1 1 1 1 1 0 1 1 1 0 22 1 1 1 1 1 0 1 1 0 0 3 0 0 0 0 0 4 4 4 12 14 38
其中 ’1’代表完好數據,’0’代表缺失值。左側第一列,’42’代表有42條數據無缺失值,第一個’9’代表9條數據Dream和NonD同時缺失。最后一行返回的就是每一個變量(列)對應的缺失數目,38為一共有多少缺失值。下圖同樣的意思。
2.2 VIM包展示數據缺失情況
1)展示sleep數據集的整體缺失情況
library("VIM")aggr(sleep,prop=FALSE,numbers=TRUE) 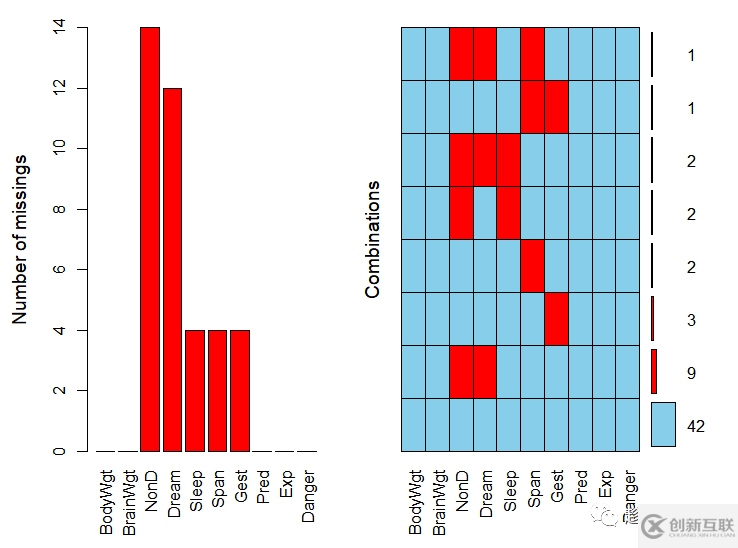
2)展示sleep數據集感興趣的變量的缺失情況
marginplot(sleep[c("Sleep","Dream")],pch=c(20),col=c("darkgray","red","blue")) 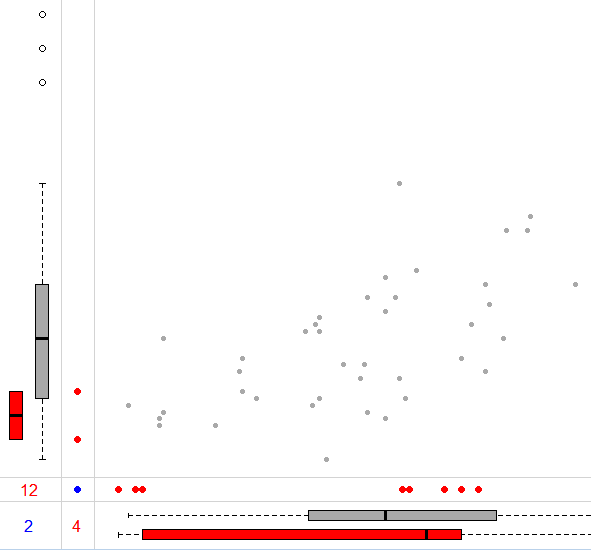
三 處理缺失值
當充分了解了缺失值的情況后,可以根據數據量的大小,以及某一列是否為重要的預測作用變量,對數據集中的NA行和某些NA列進行處理。
3.1 刪除缺失值
1)刪除數據集中所有含有NA的行和列
sleep_noNA <- na.omit(sleep)sleep_noNA <- x[complete.cases(sleep),] #兩種一樣的效果
2)刪除所有含有NA的列
na_flag <- apply(is.na(sleep), 2, sum)sleep[,which(na_flag == 0)]
3)刪除所有含有NA的行
na_flag <- apply(is.na(sleep), 1, sum)sleep[which(na_flag == 0),]
4)根據某些列的NA,移除相應的行
sleep[complete.cases(sleep[,c(1,3)]),]
4)表示將向量x中所以NA元素用某個值來代替
sleep[is.na(sleep)] <- 999
3.2 填充缺失值
當數據量不是很大或者變量比較重要時候,可以考慮對缺失值進行填充。
1)常見數值填補
library(Hmisc)data(sleep)#均值填充,適用于接近正態分布impute(sleep$NonD , mean) #中位數填充,偏態數據但是不是很嚴重impute(sleep$Dream , median)# 填充特定值impute(sleep$Span, 0)
2)DMwR包進行kNN最近鄰插補
library(DMwR)data(sleep)data <- sleep # 備份數據,對比填充結果set.seed(1120)sleep$BrainWgt[sample(nrow(sleep), 20)] <- NAhead(sleep) BodyWgt BrainWgt NonD Dream Sleep Span Gest Pred Exp Danger1 6654.000 5712.0 NA NA 3.3 38.6 645 3 5 32 1.000 6.6 6.3 2.0 8.3 4.5 42 3 1 33 3.385 NA NA NA 12.5 14.0 60 1 1 14 0.920 5.7 NA NA 16.5 NA 25 5 2 35 2547.000 4603.0 2.1 1.8 3.9 69.0 624 3 5 46 10.550 NA 9.1 0.7 9.8 27.0 180 4 4 4
# 最近鄰填補缺失值
knnOutput <- knnImputation(sleep[c(1:6)])anyNA(knnOutput)head(knnOutput)BodyWgt BrainWgt NonD Dream Sleep Span1 6654.000 5712.00000 2.534467 1.675830 3.3 38.6000002 1.000 6.60000 6.300000 2.000000 8.3 4.5000003 3.385 19.67034 10.109710 2.248604 12.5 14.0000004 0.920 5.70000 12.803345 3.353104 16.5 8.1735685 2547.000 4603.00000 2.100000 1.800000 3.9 69.0000006 10.550 95.83459 9.100000 0.700000 9.8 27.000000
# 將插補值與實際值進行對照
actuals <-data$BrainWgt[is.na(sleep$BrainWgt)]predicteds <- knnOutput[is.na(sleep$BrainWgt),"BrainWgt"]
# 兩樣本均值檢驗并計算其相似度
t.test(actuals, predicteds) # 接受差值為0的假設cor(actuals, predicteds) # 相關系數
當然根據數據和目的的不同,采用的缺失值處理方式肯定不一樣,需要我們對數據和需求有足夠的認識,做出比較好的判斷和處理。
以上就是如何進行大數據中R語言的缺失值處理,小編相信有部分知識點可能是我們日常工作會見到或用到的。希望你能通過這篇文章學到更多知識。更多詳情敬請關注創新互聯行業資訊頻道。
當前標題:如何進行大數據中R語言的缺失值處理
標題網址:http://www.yijiale78.com/article6/jcspig.html
成都網站建設公司_創新互聯,為您提供外貿網站建設、企業建站、品牌網站建設、微信小程序、、外貿建站
聲明:本網站發布的內容(圖片、視頻和文字)以用戶投稿、用戶轉載內容為主,如果涉及侵權請盡快告知,我們將會在第一時間刪除。文章觀點不代表本網站立場,如需處理請聯系客服。電話:028-86922220;郵箱:631063699@qq.com。內容未經允許不得轉載,或轉載時需注明來源: 創新互聯

- 移動網站建設如何做好極簡設計 2022-08-20
- 移動網站建設關注移動支付花樣百出,為此誰get了? 2022-07-14
- 移動網站建設體驗決定成敗 2021-12-12
- 移動網站建設怎么樣做好網站細節工作 2016-08-07
- 淺析移動網站建設的發展趨勢 2023-03-27
- 移動網站建設成功與失敗的決定性因素有哪些? 2017-01-25
- 移動網站建設與搜索引擎的關系 2016-11-07
- 分析企業移動網站建設的必要性! 2016-12-13
- 如果移動網站建設沒有注意這七點后果會是? 2022-05-27
- 未來移動網站建設成為主流,要爭取更多的流量 2023-01-04
- 移動網站建設有哪些參照標準 2021-09-03
- 深圳移動網站建設過程有哪些需要引起重視呢 2021-10-24